|
|
|
|
Curso de Doctorado
BIOINFORMATICA: Análisis
del ADN
Universidad de León.
Junio 2002.
RESULTADOS DE LA ENCUESTA FINAL A LOS ALUMNOS
![]()
VALORACIONES (1 a 10; media ±
stdev):
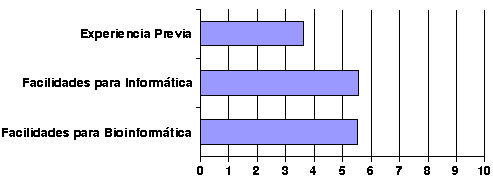
| Contexto | |||
| Experiencia previa en bioinformática: | 3.61 | ± | 2.08 |
| Facilidades en tu centro para los usuarios de informática (email, web, etc...): | 5.57 | ± | 2.33 |
| Facilidades en tu centro para los usuarios de bioinformática (GCG, web, etc...): | 5.52 | ± | 1.95 |
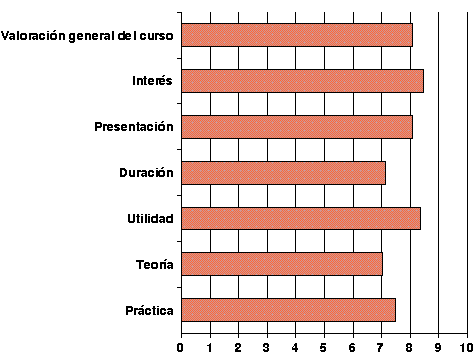
| Valoración general del curso de doctorado | |||
| Valoracion general | 8.08 | ± | 0.86 |
| Interés | 8.48 | ± | 1.38 |
| Presentación | 8.09 | ± | 1.38 |
| Duración | 7.13 | ± | 1.82 |
| Utilidad | 8.35 | ± | 1.11 |
| Teoría. | 7.04 | ± | 0.88 |
| Prácticas. | 7.48 | ± | 1.31 |
| Valoración particular de cada materia / clase: | |||
| Introducción: ¿Por qué Bioinformática?: Teoría. | 7.9 | ± | 1.33 |
| Introducción: ¿Por qué Bioinformática?: Prácticas. | 7.63 | ± | 1.21 |
| Bases de datos en Biología Molecular: Teoría. | 7.8 | ± | 1.36 |
| Bases de datos en Biología Molecular: Prácticas. | 7.68 | ± | 1.16 |
| Text queries: SRS, Entrez: Teoría | 7.95 | ± | 0.97 |
| Text queries: SRS, Entrez: Prácticas. | 7.71 | ± | 1.38 |
| Alineamiento de secuencias: Motivos, Perfiles y Dominios: Teoría. | 7.7 | ± | 1.17 |
| Alineamiento de secuencias: Motivos, Perfiles y Dominios: Prácticas. | 7.86 | ± | 1.01 |
| Analysis of DNA array data: Teoría. | 5 | ± | 2.09 |
| Analysis of DNA array data: Prácticas. | 5.61 | ± | 2.29 |
| Clustering para datos de DNA arrays: Teoría. | 5.22 | ± | 2.19 |
| Clustering para datos de DNA arrays: Prácticas. | 5.87 | ± | 2.26 |
| Detección y modelado de genes: Teoría | 8.04 | ± | 1.02 |
| Detección y modelado de genes: Prácticas. | 8.3 | ± | 0.97 |
| Análisis y Anotación de Genomas. BBDD y Comparación: Teoría. | 7.78 | ± | 1 |
| Análisis y Anotación de Genomas. BBDD y Comparación: Prácticas. | 7.96 | ± | 0.93 |
GRAFICOS:
| Contexto: |
 |
| Valoración general del curso de doctorado: |
 |
| Valoración particular de cada materia / clase: |
 |
Temas tratados que excluiría:Primers - fabricación (construcción); funcionalidad. Dibujo de árboles filogenéticos. Buena elección de temas. Análisis de secuencias escogidas por el alumno.
Lo mejor del curso:Arrays. Buena elección de temas. Lo tratado de programación: matrices... debería de tratarse en un curso distinto.
Lo peor del curso:Lo que no son arrays. Detección y modelado de genes. Las prácticas. Que está bastante orientado a la práctica y permite conocer metodologías y el funcionamiento básico de cada programa. Prácticas guiadas. Que el curso se mantenga on line. Las prácticas guiadas por los distintos programas, con una explicación de porqué es mejor utilizar uno u otro. Las prácticas orientadas y con tiempo suficiente para explorar los programas. Las páginas web. La fase práctica.
Comentario final:Arrays. DNA arrays. Teoría de DNA-arrays (demasiada teoría informática y estadística). Que ya me he olvidado de la estadística. Podría ser interesante un resumen con "recetas" prácticas para el trabajo de cada uno. A veces se pierde uno entre tantas posibilidades.
La intensidad horaria queda corta para el manejo de toda la información, además la cantidad de horas para cada tema ocasiona demasiado cansancio. Muy interesante y aplicable. Creo que mucha información para 5 días, podría ser más intensivo, y profundizar más en teoría y prácticas. Me ha gustado pero las prácticas se harían más útiles e interesantes si cada uno pudiera utilizar sus secuencias o secuencias parecidas. A veces es necesario ponerse más al nivel de los que no saben del tema...pero en general, muy buena valoración! Curso extremadamente interesante, práctico y util. Es un buen curso. El curso en general es muy interesante, salvo porque a veces se olvida un poco a quién vá dirigido. Los datos de algoritmos y programación, está bien conocer que existen, peropara alguien que a corto plazo no va a manejarlos quizás es algo prescindible. También es importante recordar que el que asiste al curso no siempre sabe, a la misma velocidad que el que lo da, donde pinchar, que link elegir... En resumen, menos matemáticas (o más lentamente explicadas) y alguna práctica echa más despacio, acabarían por mejorar un curso muy interesante. Como siempre, es demasiada densidad de datos para digerirlos en tan poco tiempo. Me ha gustado el poder seguir una práctica explicada en el texto de la página web. No he sacado conclusión alguna del tema de los arrays. Supongo que también es culpa mía.
| Protein
Design Group.
Centro Nacional de Biotecnología (CNB - CSIC). Campus Universidad Autónoma de Madrid. Cantoblanco. 28049 Madrid. Spain. Phone:+34-91-585 46 69 Fax: +34-91-585 45 06 |
Manuel J. Gómez
e-mail: mjgommo@cnb.uam.es Last update: June, 2002 |